近期,bat365在线唯一官网登录施建宇教授课题组在微生物学期刊FrontiersinMicrobiology(中科院2区Top期刊,IF=5.64)上发表了题为“NNAN: Nearest Neighbor Attention Network to predict drug-microbe associations”的生物信息交叉学科研究性论文(论文链接:https://doi.org/10.3389/fmicb.2022.846915)。bat365在线唯一官网登录博士生朱蓓及硕士生徐意为该论文的共同第一作者,施建宇教授及计算机学院于会副教授为该论文的共同通讯作者。该研究成果得到了国家自然科学基金和陕西省重点研发计划等科研项目等资助。
下文简要介绍了本文的主要内容。
1.研究背景
人体微生物群是指所有与人体有关的微生物的集合,已有的研究(包括细胞实验、动物实验、流行病学研究、临床病例等)证实了人体微生物群与人类健康有着密不可分的关系。当微生物群异常时,可能会导致多种常见的代谢紊乱疾病。比如异常肠道菌群将导致非酒精性脂肪性肝病、肥胖、糖尿病等。然而在采用口服药物治疗疾病时,许多药物可以被人体微生物代谢,药物的代谢产物会显著改变药理作用,导致患者的治疗效果降低。因此,在用药前确定潜在的药物-微生物关联(DMAs)是至关重要的。然而,由于微生物种类繁多,成本高,耗时长,传统的DMA测定方法无法在大范围内应用。因此,用计算技术预测可能的DMA是一个重要的课题。本论文设计了一个深度学习模型,命名为最近邻注意力网络(NNAN),来预测潜在DMAs。
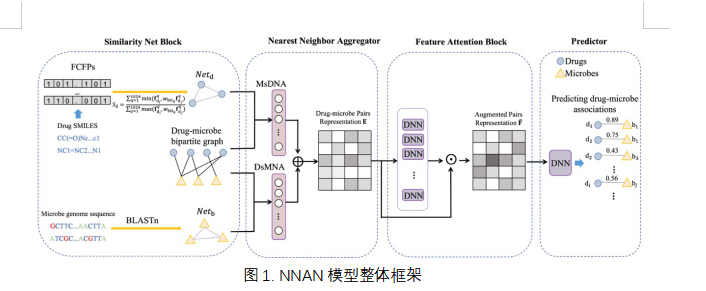
2.研究方法
通过构建“药物-微生物”关联二部图,本课题组提出了一个NearestNeighborAttentionNetwork (简称为NNAN)深度学习模型(图1),可有效应用于DMAs的相互作用预测。NNAN由四部分组成:相似性网络构建模块、近邻信息聚合器、特征注意力模块和一个预测器。相似性网络构建模块(见图1最左方)主要用于构建药物相似性网络和微生物相似性网络。近邻信息聚合器模块针对药物-微生物二部图,对网络中不同类型的实体(药物和微生物)采用基于信息传递的机制,聚合不同实体邻居的信息,分别生成药物和微生物的嵌入表示,并将药物嵌入表示和微生物嵌入表示拼接,得到“药物-微生物对”嵌入特征。特征注意力模块将“药物-微生物”嵌入特征输入至一组常规深度神经网络中,生成了一个关于“药物-微生物”嵌入特征的注意力矩阵,且将该注意力矩阵与原“药物-微生物”嵌入特征进行元素积运算,最终得到一个特征重要性选择后的矩阵。最后,我们利用一个全连接深度神经网络作为一个二部分类器,预测潜在DMAs。
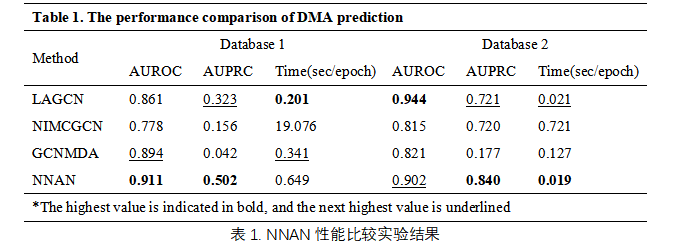
为评估NNAN模型性能,我们使用5折交叉验证法在两个公开数据集上与三种基线方法进行比较实验,并使用AUROC与AUPRC作为衡量预测潜在DMAs的性能指标,验证分子结构相似的药物是否可以与基因序列相似的微生物发生相互作用。综合结果表明,NNAN优于其他竞争方法(表1)。
为验证NNAN模型有效性,我们实施了一个实例分析:验证NNAN是否能找出微生物Bacteroides fragilis和其他药物之间的潜在联系。我们提取了邻接矩阵中标记为零的DMAs(即数据集中表示为未知/不可发生相互作用),并根据DMAs预测分数将其降序排列。我们通过文献检索验证前10、20和50个预测的候选药物来评估NNAN的性能。最终结果表明,在预测的前10、20和50个药物中,分别有10个、17个和38个药物被以前发表的文献证实其可以与Bacteroides fragilis发生作用。上述结果再次证明,NNAN是一个有效的DMA预测模型。
Citations:Zhu B, Xu Y, Zhao P, Yiu S-M, Yu H and Shi J-Y* (2022) NNAN: Nearest Neighbor Attention Network to Predict Drug–Microbe Associations.Front. Microbiol.13:846915. doi: 10.3389/fmicb.2022.84691(文/图:朱蓓;审核:杨慧)